
生命体的细胞功能多样性源于细胞的异质性和组织环境的复杂性,解析基因表达的时空模式有助于理解细胞间相互作用、组织的微环境调控、动态调控模式等重要方面,对揭示生命活动(如胚胎发生、特定脑功能)机理和疾病(如肿瘤、神经精神疾病)发生机制等核心生物学问题至关重要。近年来,单细胞RNA测序(scRNA-seq)技术促进了组织细胞异质性的解析,但是缺乏组织微环境和细胞空间信息,限制了对生命信息的深度解读。空间组学技术通过解析生物分子的表达水平和空间位置,极大地促进了细胞中基因空间表达谱的探究,加速了我们对组织微环境和功能机制的深入理解。2022年,空间组学技术被国际学术期刊《Nature》评为年度七大颠覆性技术;2023年,世界经济论坛发布《2023年十大新兴技术报告》,空间组学被评为最有潜力、对世界产生积极影响的十大技术之一。
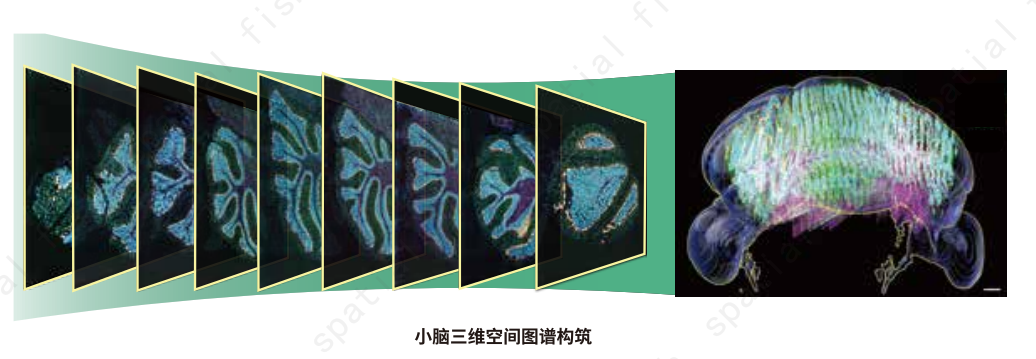
生物体本质上是三维的,了解复杂的生物系统需要在三维背景下对组织器官甚至全身进行分析,才能更精准地探寻生命的机理。3D空间组学将从更完整、更全面的角度展现组织块、器官甚至整个生命体的细胞与分子空间构筑模式,助力器官功能、胚胎发育、癌症等复杂机制以及物种演化等重要科学问题的阐明。
目前市场上的空间组学主要是在组织切片展现基因表达的2D空间组,对于3D水平的空间组检测还十分缺乏。为了满足日益增多的3D空间组检测需求,鲲羽生物推出两种3D空间组检测方案:一种是将组织块、器官等样本进行连续或半连续切片,将每张切片进行2D空间组学检测,最终将所有多张切片的2D成像数据对准后实现3D重构,实现3D空间组学检测;第二种是基于厚组织、器官或生命体的直接透明与空间组学检测结合,通过3D扫描成像获取X轴、Y轴和Z轴的单细胞精度基因表达谱空间信息,实现真正的三维空间组检测。
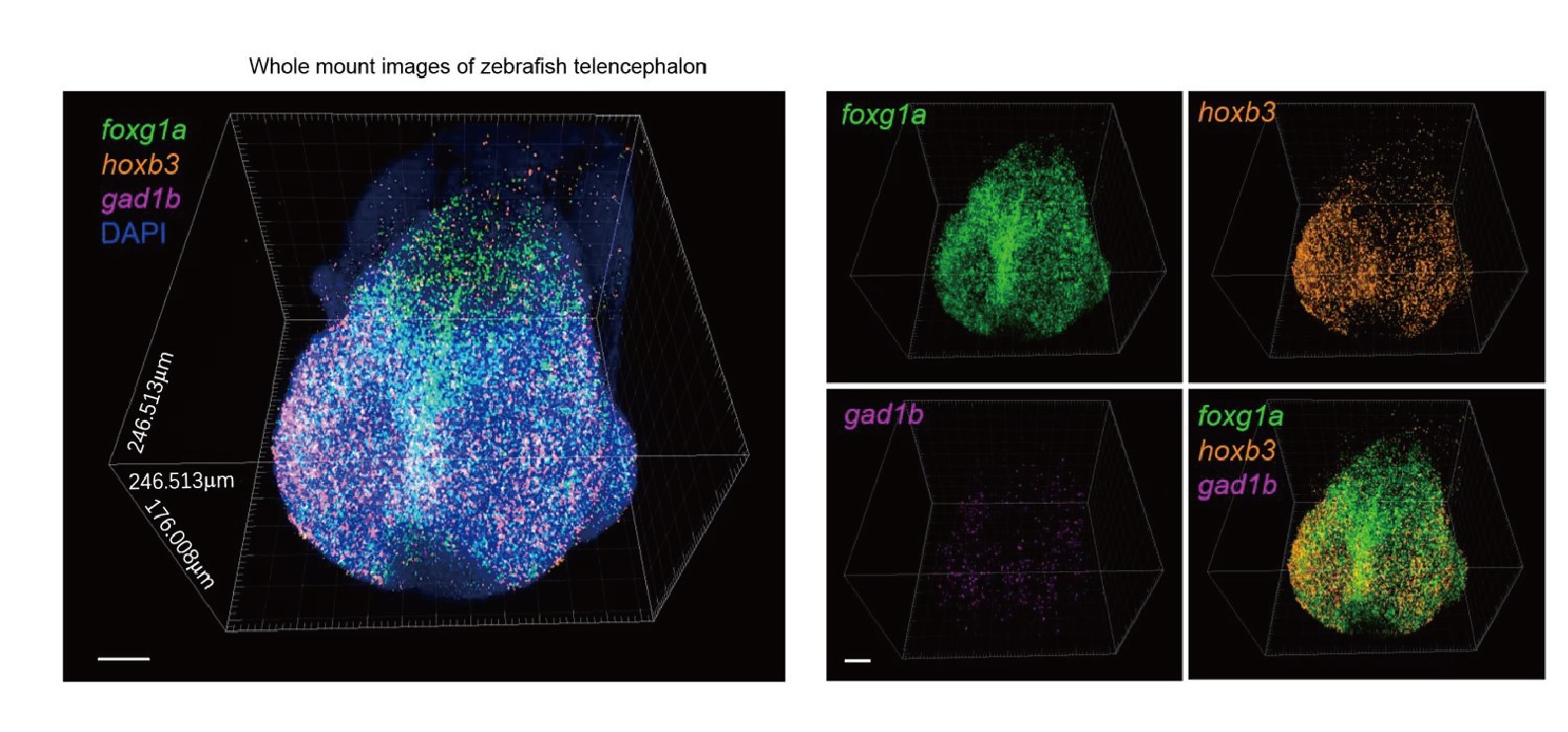
斑马鱼端脑三维空间图谱构筑
鲲羽生物为您提供的3D空间组学检测实现了三维、高空间分辨率、特异性和高通量的基因表达谱解析,为构建精确的、全面的组织3D图谱提供了必要支持,必将为您的科研和临床需求提供重要保障,也将大大推动我们对于生命复杂性和人类疾病的全面认知。
参考文献
1. Asp M, Giacomello S, Larsson L, et al. A Spatiotemporal Organ-Wide Gene Expression and Cell Atlas of the Developing Human Heart. Cell. 2019 Dec 12;179(7):1647-1660.e19.
2. Waylen, L.N., Nim, H.T., Martelotto, L.G. et al. From whole-mount to single-cell spatial assessment of gene expression in 3D. Commun Biol 3, 602 (2020).
3. Ao Chen, Sha Liao, Mengnan Cheng, et al. Spatiotemporal transcriptomic atlas of mouse organogenesis using DNA nanoball-patterned arrays. Cell, 2022, 185(10): 1777-1792.e21.
4. Xiaoyu Wei, Sulei Fu, Hanbo Li, et al. Single-cell Stereo-seq reveals induced progenitor cells involved in axolotl brain regeneration. Science, 2022, 377(6610): eabp9444.
5. Mingyue Wang, Qinan Hu, Tianhang Lv, et al. High-resolution 3D spatiotemporal transcriptomic maps of developing Drosophila embryos and larvae. Developmental Cell, 2022, 57(10): 1271-1283.e4.
6. Liao J, Lu X, Shao X, et al. Uncovering an Organ's Molecular Architecture at Single-Cell Resolution by Spatially Resolved Transcriptomics. Trends Biotechnol. 2021 Jan;39(1):43-58.